데이콘(생육 환경 최적화 경진대회) : 중복 데이터
최종결과 : 중복 데이터
중복 데이터1
#### 1. 분석 준비 ####
pacman::p_load(ggpmisc, magick, imager, tidyverse) # 데이터 전처리 관련 패키지
#### 2. 기본 데이터 로딩 ####
train_labels <- list.files(path="label", full.names = TRUE) %>%
lapply(read_csv, show_col_types = F) %>% bind_rows()
#### 3. 중복데이터 ####
#### 가. 완전히 같은 데이터 ####
images1 <- function(case="CASE59", num=c("10", "20", "30")){
ggsave(filename = "data/temp.png", # 임시로 그래프를 이미지로 저장
train_labels %>%
mutate(num=as.integer(str_sub(img_name, 8, 9))) %>%
filter(grepl(case, img_name)) %>%
ggplot(aes(num, leaf_weight)) +
geom_line(size=0.4) + theme_bw()+
theme(text = element_text(size=3.5),
plot.margin = margin(0, 0, 0.1, 0.1, "cm"),
# top, right, bottom, left (사진과의 적당한 여백 유지)
panel.border = element_rect(size = 0.3),
panel.grid = element_line(size=0.2),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
axis.ticks = element_line(size = 0.2),
axis.ticks.length = unit(.03, "cm")) +
labs(x=""),
width = 200, height = 150, dpi = 300, units = "px", device='png')
img1_name <- train_labels %>% mutate(img_name=paste0("train/", img_name)) %>%
filter(grepl(paste0(case, "_", num[1]), img_name))
img1 <- image_read(img1_name$img_name) %>% image_scale(200) %>%
image_annotate(paste0(str_sub(img1_name, 7, -1), ", ", round(img1_name$leaf_weight)),
size=12, color="blue", boxcolor="white", location="+40+135")
img2 <- image_read("data/temp.png")
img12 <- image_append(c(img1, img2))
img3_name <- train_labels %>% mutate(img_name=paste0("train/", img_name)) %>%
filter(grepl(paste0(case, "_", num[2]), img_name))
img3 <- image_read(img3_name$img_name) %>% image_scale(200) %>%
image_annotate(paste0(str_sub(img3_name, 7, -1), ", ", round(img3_name$leaf_weight)),
size=12, color="blue", boxcolor="white", location="+40+135")
img4_name <- train_labels %>% mutate(img_name=paste0("train/", img_name)) %>%
filter(grepl(paste0(case, "_", num[3]), img_name))
img4 <- image_read(img4_name$img_name) %>% image_scale(200) %>%
image_annotate(paste0(str_sub(img4_name, 7, -1), ", ", round(img4_name$leaf_weight)),
size=12, color="blue", boxcolor="white", location="+40+135")
img34 <- image_append(c(img3, img4))
image_append(c(img12, img34), stack=T)
}
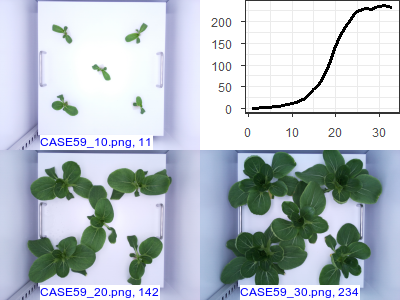
images1("CASE59", c(10, 20, 30))
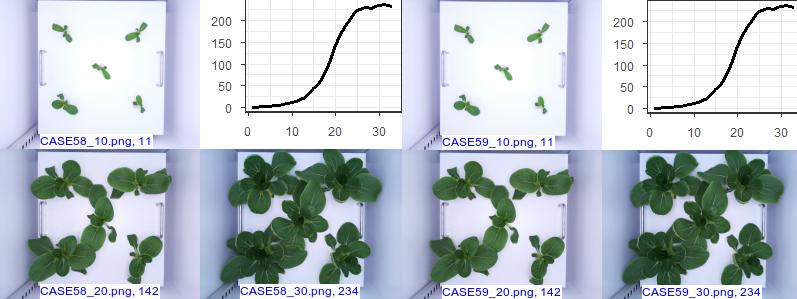
images1("CASE58", c(10, 20, 30))
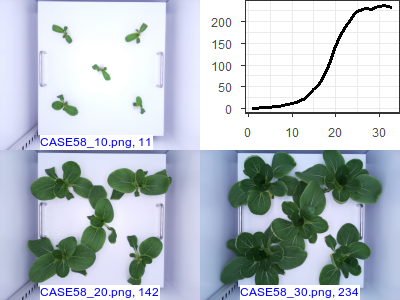
CASE59와 CASE58이 완벽하게 같은 데이터 임을 알 수 있습니다. CASE59의 경우 메타데이터가 결측치이므로 CASE59 전체를 삭제하는 것이 바람직합니다.
중복데이터2
images1("CASE10", c(32, 33, 34))
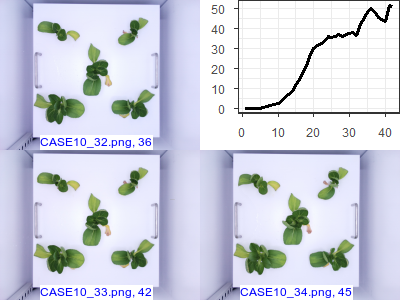
images1("CASE11", c(32, 33, 34))
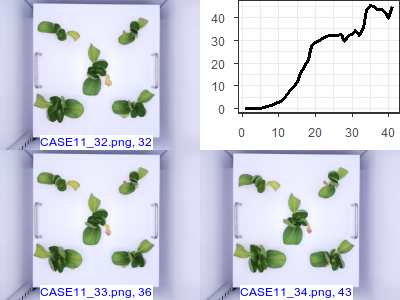
images1("CASE12", c(32, 33, 34))
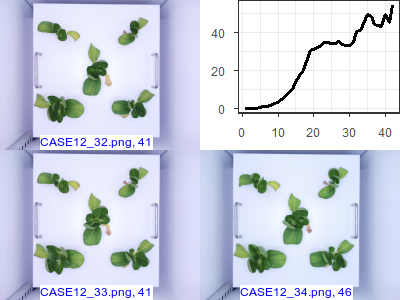
images1("CASE13", c(32, 33, 34))
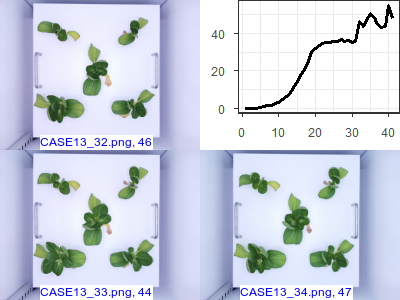
images1("CASE14", c(32, 33, 34))
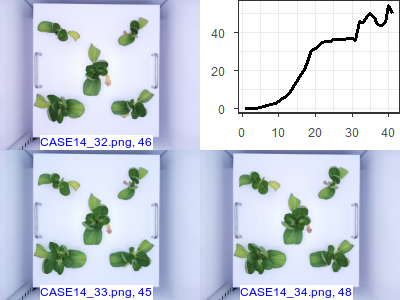
CASE10~CASE14의 패턴을 보면 대동 소이합니다. 메타 데이터를 살펴보면 하나의 청경채를 가지고 부풀려진 데이터임을 알 수 있습니다.
CASE10은 2021-06-02 19:59 분부터 시작해서 2021-07-14 19:58 분에 끝납니다.
CASE11은 2021-06-03 02:46 분부터 시작해서 2021-07-14 02:45 분에 끝납니다.
CASE12는 2021-06-03 08:59 분부터 시작해서 2021-07-15 08:58 분에 끝납니다.
CASE13은 2021-06-03 11:59 분부터 시작해서 2021-07-14 11:58 분에 끝납니다.
CASE14는 2021-06-03 16:59 분부터 시작해서 2021-07-14 16:58 분에 끝납니다.
성장 패턴이 동일하기 때문에 같은 청경채라고 의심할 수 있는데 날짜까지 겹치는 것으로 보아 동일한 청경채임이 확실합니다. 3~6시간 정도의 간격을 두 촬영한 데이터입니다. 이러한 이상치는 청경채의 일반적인 특성이 아닌 독특한 청경채 하나의 케이스에 치우치는 모델을 만들 수 있기 때문에 제거하는 것이 바람직합니다. 하지만 완전히 동일한 데이터는 아니기 때문에 경우에 따라서는 사용할 수도 있을 것 같습니다. 지금까지는 CASE14를 제외하고 모두 제거한 후 분석했는데 CASE10~CASE14까지 하나의 그룹으로 분류되어 다른 데이터에 영향을 미치지 않는다는 사실을 알고난 후 제거했던 CASE10~CASE13을 모두 넣어서 분석해 보려고 생각중입니다.
특이한 점은 32번째부터 41, 42번째까지는 무게가 들쭉 날쭉하는 것을 확인할 수 있습니다. 잎이 시들면서 시든 잎을 제외한 잎의 무게를 추정하면서 생긴 오차라고 생각됩니다. 이 부분을 제거할지 포함시킬지는 분석자가 판단할 몫입니다. 이 글을 쓰기 전까지는 분석에서 제외했는데 시든 잎의 사례가 적은 만큼 넣어서 분석하는 것도 타당한 것 같기도 합니다. 해당 데이터(사진, 혹은 메타 데이터)를 추가해서 리더보드에 올려 봐야할 것 같습니다.
댓글남기기