데이콘(생육 환경 최적화 경진대회) : 이미지 전처리2
최종 결과물 확인
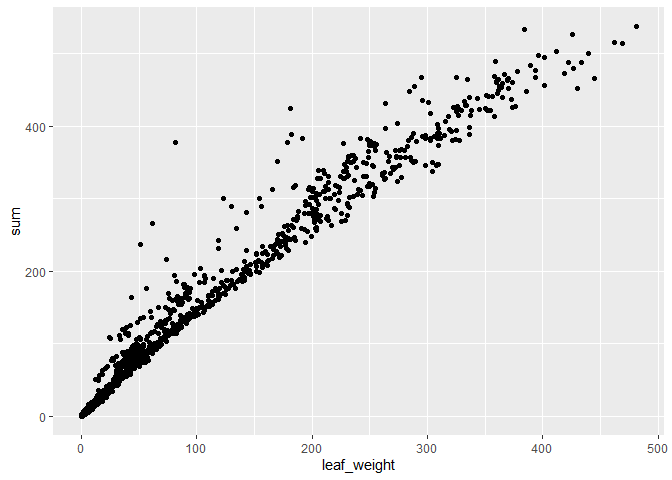 이미지 마스킹을 해서 픽셀값을 모두 합한 값(sum)과 실제 잎의 무게(leaf_weight)의 관계입니다.
이미지 마스킹을 해서 픽셀값을 모두 합한 값(sum)과 실제 잎의 무게(leaf_weight)의 관계입니다.
이미지 오리기
#### 1. 분석 준비 ####
pacman::p_load(imager, magick, tidyverse) # 데이터 전처리 관련 패키지
img_train <- list.files("train", full.names = T) # train 폴더 속 이미지 이름
img_test <- list.files("test", full.names = T) # test 폴더 속 이미지 이름
#### 2. 이미지 오리기 ####
img <- image_read('train/CASE07_02.jpg') %>% image_scale("328x246") # magick 패키지 활용
paste1 <- image_crop(img, "15x15+165+100") # 청경채
paste2 <- image_crop(img, "75x20+10+130") %>% image_rotate(90) # 손잡이
paste3 <- image_crop(img, "20x20+140+215") # 경계
paste4 <- image_crop(img, "20x20+62+217") # 철제 프레임
img <- load.image('train/CASE07_02.jpg') %>% resize(328, 246) # imager 패키지 활용
px.fg <- ((Xc(img) %inr% c(165, 180)) & (Yc(img) %inr% c(100, 115))) # 청경채
px.bg1 <- ((Xc(img) %inr% c(10, 85)) & (Yc(img) %inr% c(130,150))) # 손잡이
px.bg2 <- ((Xc(img) %inr% c(140, 160)) & (Yc(img) %inr% c(215, 235))) # 경계
px.bg3 <- ((Xc(img) %inr% c(62, 82)) & (Yc(img) %inr% c(217, 237))) # 철제 프레임
img %>% plot()
highlight(px.fg)
highlight(px.bg1,col="blue")
highlight(px.bg2,col="blue")
highlight(px.bg3,col="blue")

빨간색 부분이 청경채를 구분할 샘플 이미지입니다.
파란색으로 된 손잡이, 박스 경계, 철제 프레임은 배경으로 처리할 예정입니다.
masking
ez_seg <- function(file="train/CASE07_02.jpg", p1=paste1, p2=paste2, k=1){
img <- image_read(file) %>% image_scale("328x246")
img <- image_composite(img, paste1, offset="+0+0") %>% # 청경채 붙이기
image_composite(paste2, offset="+308+80") %>% # 손잡이 붙이기
image_composite(paste3, offset="+308+80") %>% # 박스 경계 붙이기
image_composite(paste4, offset="+308+100") # 철제 프레임 붙이기
img <- magick2cimg(img) # imager 데이터로 변경
px.fg <- ((Xc(img) %inr% c(0, 15)) & (Yc(img) %inr% c(0, 15))) # 청경채 좌표
px.bg <- ((Xc(img) %inr% c(308, 328)) & (Yc(img) %inr% c(80,155)))# 배경들 좌표
im.lab <- sRGBtoLab(img) # RGB를 CIELAB로
cvt.mat <- function(px) matrix(im.lab[px], sum(px)/3, 3) # 이하코드는 잘...
fgMat <- cvt.mat(px.fg)
bgMat <- cvt.mat(px.bg)
labels <- c(rep(1, nrow(fgMat)), rep(0, nrow(bgMat)))
testMat <- cvt.mat(px.all(img))
out <- nabor::knn(rbind(fgMat, bgMat), testMat, k=k) # knn 적용
out <- labels[as.vector(out$nn.idx)] %>% matrix(dim(out$nn.idx)) %>% rowMeans
msk <- as.cimg(rep(out, 3), dim=dim(img)) # 마스크로 만들기
return(msk)
}
ez_check1 <- function(name="train/CASE01_01.png", k=200){
img1 <- load.image(name) %>% resize(328, 246)
img2 <- ez_seg(name, k=k)
result <- list(img1, img2) %>% imappend("x") %>% plot()
}
ez_check2 <- function(name="train/CASE01_01.png", k=200, x=33, y=25){
img1 <- image_read(name) %>% image_scale("328x246") %>% magick2cimg()
img2 <- ez_seg(name, k=k)
img3 <- img2 %>% resize(x,y)
dxy <- 3
img4 <- img2 %>% resize(x,y) %>% as.data.frame() %>%
mutate(value=ifelse(x<dxy & y<dxy, 0, value)) %>%
filter(cc==1) %>% pull(value) %>% as.cimg(x=x, y=y)
result <- list(img3, img4) %>% imappend("x") %>% plot()
}
데이콘에 샘플로 공개된 이미지가 3~4M를 초과할 정도로 용량이 매우 큰 편입니다. 3280*2464의 해상도를 가지고 있는데 이를 1/10 수준으로 줄여서 배경과 청경채를 붙여 넣습니다. 이 과정은 magick 패키지를 이용하였습니다. 일단 이미지를 불러오는 데 걸리는 시간이 짧고 크기가 다른 이미지를 합칠 수 있는 장점도 가지고 있습니다.
청경채와 배경을 구분하는 코드를 적용하기 위해 magick 파일을 imager에서 사용할 수 있는 형태로 바꾸었습니다. 나머지 코드는 https://dahtah.github.io/imager/foreground_background.html를 참고했습니다.
ez_check1() 함수는 원본 사진과 마스킹한 결과를 보여주고, ez_check2() 함수는 마스킹한 결과를 다시 33*25 사이즈로 줄이고 청경채를 구분하기 위해 0,0 좌표에 붙였던 청경채를 잘라낸 후 첫번째 채널의 데이터를 보여줍니다.
적용 결과 확인하기1
ez_check1("train/CASE05_21.png")

ez_check2("train/CASE05_21.png", x=33, y=25)
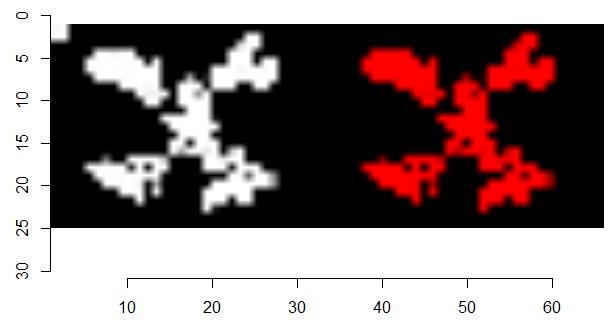 왼쪽 위에 있는 흰색 사각형이 오른쪽 그림에서는 없어진 것을 확인할 수
있습니다. 오른쪽 그림은 첫번째 채널의 데이터만 남겼기 때문에 붉게 보이는
것입니다.
왼쪽 위에 있는 흰색 사각형이 오른쪽 그림에서는 없어진 것을 확인할 수
있습니다. 오른쪽 그림은 첫번째 채널의 데이터만 남겼기 때문에 붉게 보이는
것입니다.
적용 결과 확인하기2
ez_check1(img_train[35], k=200)

ez_check2(img_train[35], x=33, y=25)
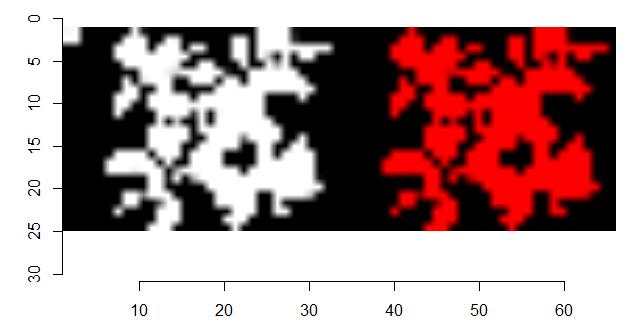
적용 결과 확인하기3
ez_check1(img_train[20], k=200)
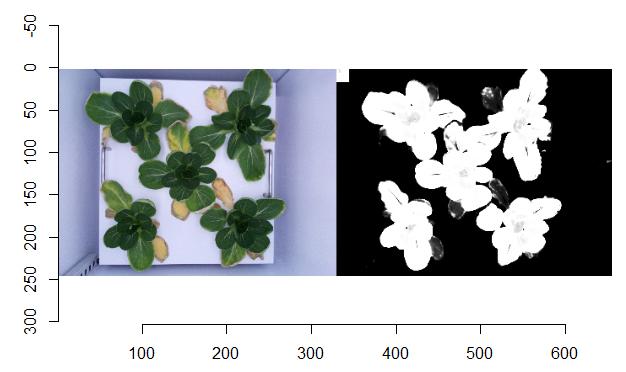
ez_check2(img_train[20], x=33, y=25)
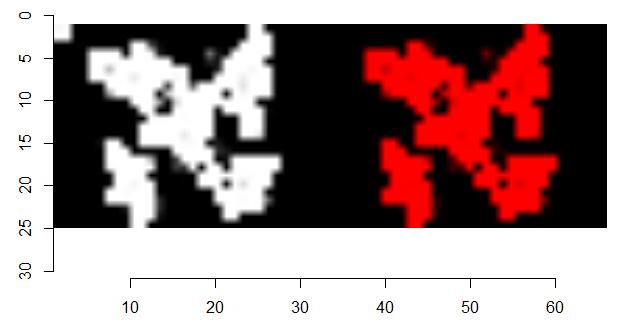 누렇게 뜬 잎을 잘 구분해서 삭제해 줍니다. k를 200으로 설정해 주어서 이렇게 된
것입니다. k를 1로 맞추면 누렇게 뜬 잎도 정상적인 잎처럼 인식을 하게 만들
수도 있습니다. 이 대회에서의 잎 면적(질량)은 누렇게 뜬 잎을 제외한
값이라고 하므로 k를 200 정도로 맞추었습니다.
누렇게 뜬 잎을 잘 구분해서 삭제해 줍니다. k를 200으로 설정해 주어서 이렇게 된
것입니다. k를 1로 맞추면 누렇게 뜬 잎도 정상적인 잎처럼 인식을 하게 만들
수도 있습니다. 이 대회에서의 잎 면적(질량)은 누렇게 뜬 잎을 제외한
값이라고 하므로 k를 200 정도로 맞추었습니다.
적용 결과 확인하기4
ez_check1(img_train[115], k=200)

ez_check2(img_train[115], x=33, y=25)
 잎의
면적이 가장 큰 것을 테스트 해봤습니다. 보이지 않아야할 구멍들이 보이긴
합니다. k를 1로 맞추면 잎이 꽉 차게 나옵니다. 주최측에 문의한 결과
눈으로 보고 특정 조건에 해당할 경우 설정을 바꾸면 안된다고 해서 일관되게
k를 200으로 맞추었습니다. 나중에 확인해보니 이 정도의 차이는 큰 차이가
아니었습니다.
잎의
면적이 가장 큰 것을 테스트 해봤습니다. 보이지 않아야할 구멍들이 보이긴
합니다. k를 1로 맞추면 잎이 꽉 차게 나옵니다. 주최측에 문의한 결과
눈으로 보고 특정 조건에 해당할 경우 설정을 바꾸면 안된다고 해서 일관되게
k를 200으로 맞추었습니다. 나중에 확인해보니 이 정도의 차이는 큰 차이가
아니었습니다.
적용 결과 확인하기5
ez_check1(img_train[473], k=200)
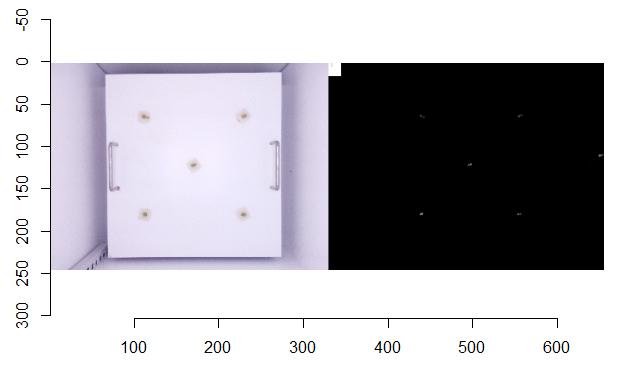
ez_check2(img_train[473], x=33, y=25)
 잎의
면적이 0인 것을 테스트 해봤습니다. 조그마한 점들이 보이는데 청경채를
심은 틀을 청경채로 오인한 것 같습니다. 조그마한 틀 이미지도 배경으로
추가하면 이것도 제거할 수 있습니다.
잎의
면적이 0인 것을 테스트 해봤습니다. 조그마한 점들이 보이는데 청경채를
심은 틀을 청경채로 오인한 것 같습니다. 조그마한 틀 이미지도 배경으로
추가하면 이것도 제거할 수 있습니다.
일괄 적용하기
rdata <- NULL
for(i in img_train){
rdata <- rdata %>%
bind_rows(ez_seg(i, k=200) %>% resize(33, 25) %>% # 사이즈 줄이기
as.data.frame() %>% filter(cc==1) %>% # 1번째 채널만 선택
mutate(value=ifelse(x<3 & y<3, 0, value)) %>% # 왼목 모서리 제거
rownames_to_column("p") %>% # 데이터 index
mutate(p=paste0("p", p)) %>% select(p, value) %>% # 변수명으로 치환
pivot_wider(names_from="p", values_from="value") %>% # wide form으로
mutate(img_name=i, .before=1)) # 이미지 이름 포함
}# 컴퓨터 성능에 따라 1시간 가까이 걸릴 수도 있습니다.
rdata %>% mutate(img_name=str_remove(img_name, "train/")) %>%
write.csv("rdata_200.csv", row.names=F) # 결과 저장
for(i in img_test){
test <- test %>%
bind_rows(ez_seg(i, k=200) %>% resize(33, 25) %>%
as.data.frame() %>% filter(cc==1) %>%
mutate(value=ifelse(x<3 & y<3, 0, value)) %>%
rownames_to_column("p") %>%
mutate(p=paste0("p", p)) %>% select(p, value) %>%
pivot_wider(names_from = "p", values_from = "value") %>%
mutate(img_name=i, .before=1))
}
test %>% mutate(img_name=str_remove(img_name, "test/")) %>%
write.csv("test_200.csv", row.names=F)
labels <- list.files(path="label", full.names = TRUE) %>%
lapply(read_csv, show_col_types = F) %>% bind_rows
rdata <- labels %>% left_join(rdata) %>%
mutate(sum=rowSums(select(., p1:p825)), .before=3) %>%
separate(img_name, into=c("img_name", "num"), sep="\\.") %>% select(-num)
rdata %>% ggplot(aes(leaf_weight, sum)) + geom_point()
 실제 무게와 상당히 유사한 결과를 확인할 수 있습니다. 일부 이미지(CASE73_10,
CASE23_1~8) 등이 실제 무게보다 더 크게 값이 인식되긴 했지만 이 정도는
무난해 보입니다.
실제 무게와 상당히 유사한 결과를 확인할 수 있습니다. 일부 이미지(CASE73_10,
CASE23_1~8) 등이 실제 무게보다 더 크게 값이 인식되긴 했지만 이 정도는
무난해 보입니다.
이렇게 얻어진 데이터를 AutoML로 학습시켜서 test 데이터에 적용해서 리더보드에 올리면 0.25~0.30 정도 나오는 것 같습니다. 오로지 이미지만 가지고 얻은 결과이고 AutoML의 한계 때문에 좋은 수준의 성적은 아닙니다.
댓글남기기